RT-PCR引物设计的方法步骤
RT-PCR引物设计的方法步骤如下:
1、在 NCBI 上搜索到该基因,找到该基因的 mRNA ,在 CDS 选项中,找到编码区所在位置,在下面的origin 中, Copy 该编码序列作为软件查询序列的候选对象。
2、打开 Primer Premier5 ,点击 File-New-DNA sequence, 出现输入序列窗口, Copy 目的序列在输入框内(选择 As),此窗口内,序列也可以直接翻译成蛋白。点击 Primer ,进入引物窗口。
3、此窗口可以链接到 “引物搜索 ”、 “引物编辑 ”以及 “搜索结果 ”选项,点击 Search 按钮,进入引物搜索框,选择 “PCR primers ”, “Pairs,”设定搜索区域和引物长度和产物长度。在 Search Parameters 里面,可以设定相应参数。一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100 ~ 200bp 的产物电泳跑得较散,所以可以选择 300 ~ 500bp 。
4、点击 OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating )排序,点击其中任一个搜索结果,可以在 “引物窗口 ”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
5、对于引物的序列,可以简单查看一下,避免出现下列情况: 3’端不要以 A 结尾,好是 G 或者 C,T 也可以; 3’不要出现连续的 3 个碱基相连的情况,比如 GGG 或 CCC,否则容易引起错配。此窗口中需要着重查看的包括: Tm 应该在 55 ~ 70 度之间, GC%应该在 45 %~ 55 %间,上游引物和下游引物的 Tm 值好不要相差太多,大概在 2 度以下较好。该窗口的下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为 “None”为好。但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。对于引物具体详细的评价需要借助于 Oligo 来完成, Oligo 自身虽然带有引物搜索功能, 但其搜索出的引物质量感觉不如 Primer5 。
6、在 Primer5 窗口中,若觉得某一对引物合适,可以在搜索结果窗口中,点击该引物,然后在菜单栏,选择 File-Print-Current pair ,使用 PDF 虚拟打印机,即可转换为 Pdf 文档,里面有该引物的详细信息。
7、在 Oligo 软件界面, File 菜单下,选择 Open ,定位到目的 cDNA 序列(在 primer 中,该序列已经 被保存为 Seq 文件),会跳出来两个窗口,分别为 Internal Stability ( Delta G )窗口和 Tm 窗口。在 Tm 窗口中,点击左下角的按钮,会出来引物定位对话框,输入候选的上游引物序列位置(Primer5 已经给出)即可,而引物长度可以通过点击 Change - Current oligo length 来改变。定位后,点击 Tm 窗口的 Upper 按钮,确定上游引物,同样方法定位下游引物位置,点击 Lower 按钮, 确定下游引物。引物确定后,即可以充分利用 Analyze 菜单中各种强大的引物分析功能了。
8、Analyze 中,第一项为 Key info ,点击 Selected primers ,会给出两条引物的概括性信息,其中包括引物的 Tm 值,此值 Oligo 是采用 nearest neighbor method 计算,会比 Primer5 中引物的 Tm 值略高,此窗口中还给出引物的 Delta G 和 3’端的 Delta G 。3’端的 Delta G 过高,会在错配位点形成双 链结构并引起 DNA 聚合反应,因此此项绝对值应该小一些,好不要超过 9。
9、Analyze 中第二项为 Duplex Formation ,即二聚体形成分析,可以选择上游引物或下游引物,分析上游引物间二聚体形成情况和下游引物间的二聚体情况,还可以选择 Upper/Lower ,即上下游引物之间的二聚体形成情况。引物二聚体是影响 PCR 反应异常的重要因素,因此应该避免设计的引物存在二聚体,至少也要使设计的引物形成的二聚体是不稳定的,即其 Delta G 值应该偏低,一般不要使其超过 4.5kcal/mol ,结合碱基对不要超过 3 个。 Oligo 此项的分析窗口中分别给出了 3’端和整个引物的二聚体图示和 Delta G 值。
10、Analyze 中第三项为 Hairpin Formation ,即发夹结构分析。可以选择上游或者下游引物,同样,Delta G 值不要超过 4.5kcal/mol ,碱基对不要超过 3 个。
11、Analyze 中第四项为 Composition and Tm ,会给出上游引物、下游引物和产物的各个碱基的组成比例和 Tm 值。上下游引物的 GC%需要控制在 40 %~ 60 %,而且上下游引物之间的 GC%不要相差太大。 Tm 值共有 3 个,分别采用三种方法计算出来,包括 nearest neighbor method 、% GCmethod 和 2(A + T) + 4(G + C)method ,后一种应该是 Primer5 所采用的方法, Tm 值可以控制在50 ~ 70 度之间。
12、第五项为 False Priming Sites ,即错误引发位点,在 Primer5 中虽然也有 False priming 分析,但不如 oligo 详细,并且 oligo 会给我正确引发效率和错误引发效率,一般的原则要使错误引发效率在100 以下,当然有时候正确位点的引发效率很高的话,比如达到 400 ~ 500 ,错误引发效率超过100 幅度若不大的话,也可以接受。
13、Analyze 中,有参考价值的后一项是 “ PCR,”在此窗口中,是基于此对引物的 PCR 反应 Summary ,并且给出了此反应的佳退火温度,另外,提供了对于此对引物的简短评价。若该引物有不利于PCR 反应的二级结构存在,并且 Delta G 值偏大的话, Oligo 在后的评价中会注明,若没有注明此项,表明二级结构能值较小,基本可以接受。
14、引物评价完毕后,可以选择 File- Print ,打印为 PDF 文件保存,文件中将会包括所有 Oligo 软件中已经打开的窗口所包括的信息,多达数页。因此,打印前好关掉 Tm 窗口和 Delta G 窗口,可以保留引物信息窗口、二级结构分析窗口(若存在可疑的异常的话)和 PCR 窗口。
15、引物确定后,对于上游和下游引物分别进行 Blast 分析,一般来说,多少都会找到一些其他基因的同源序列,此时,可以对上游引物和下游引物的 blast 结果进行对比分析,只要没有交叉的其他基因的同源序列就可以。
-
论文打印要求是什么,单面还是双面? 132188
-
ieee论文什么水平,含金量如何? 71432
-
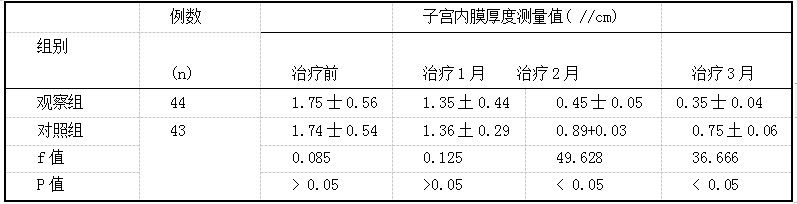
左炔诺孕酮不同给药途径对患者子宫内膜增生症的临床疗效评价 2019.10.30 15:05
-
胸腺肽与恩替卡韦联用对患者原发性肝癌的临床疗效及其对预后的影响 2019.10.30 14:47
-
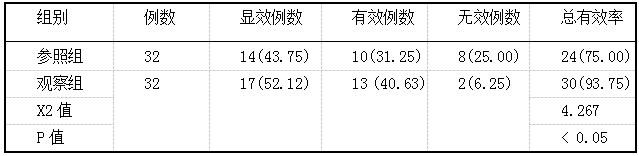
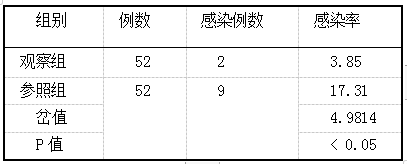
甲硝唑片官腔置入与左氧氟沙星口服对人工流产患者术后抗感染的临床疗效评价 2019.10.30 14:28
-
瑞舒伐他汀与常规降压药物联用对反勺型高血压患者降压疗效的影响 2019.10.30 11:35
-
 DNA的琼脂糖凝胶电泳实验原理和步骤
DNA的琼脂糖凝胶电泳实验原理和步骤 -
提取RNA及用DEPC去除RNase的实验经验
-
 医学生物学电子显微镜图谱
医学生物学电子显微镜图谱 -
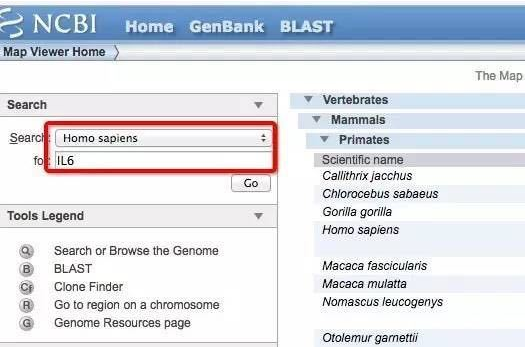 如何使用 NCBI 查找基因序列、mRNA和Promoter
如何使用 NCBI 查找基因序列、mRNA和Promoter